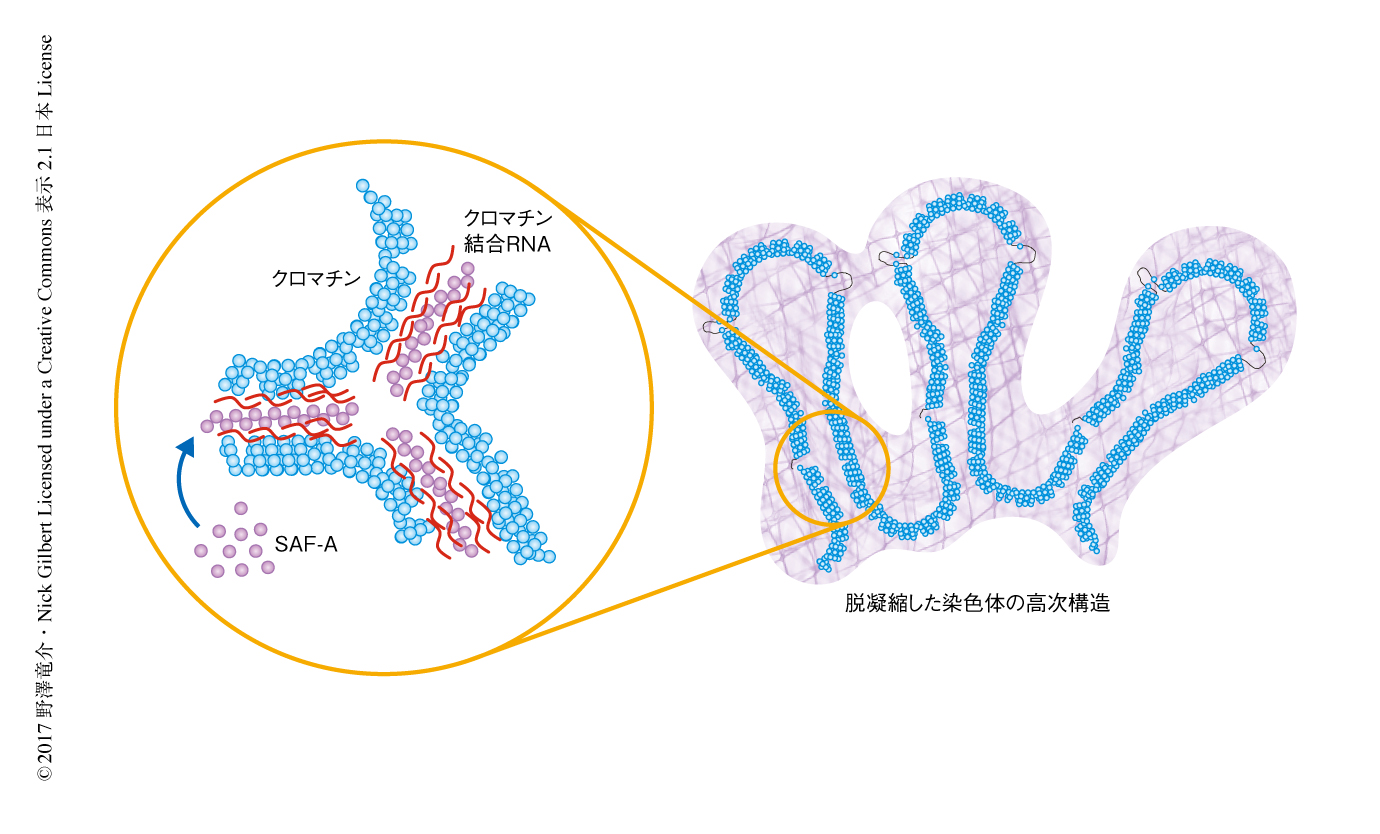
SAF-Aはオリゴマーを形成しクロマチン結合RNAとともに染色体の構造を制御する
野澤竜介・Nick Gilbert
(英国Edinburgh大学Institute of Genetics and Molecular Medicine)
email:野澤竜介
DOI: 10.7875/first.author.2017.068
SAF-A regulates interphase chromosome structure through oligomerization with chromatin-associated RNAs.
Ryu-Suke Nozawa, Lora Boteva, Dinesh C. Soares, Catherine Naughton, Alison R. Dun, Adam Buckle, Bernard Ramsahoye, Peter C. Bruton, Rebecca S. Saleeb, Maria Arnedo, Bill Hill, Rory R. Duncan, Sutherland K. Maciver, Nick Gilbert
Cell, 169, 1214-1227.e18 (2017)
真核生物においてDNAはクロマチン構造をとり密に折りたたまれて核に収納されている.しかし,その染色体の構造を維持あるいは制御する分子機構の全容はいまだ明らかにされていない.筆者らは,SAF-Aがクロマチン結合RNAと結合し,ヒトの間期における染色体の高次構造を転写に依存的に制御することを見い出した.その分子機構についてくわしく調べたところ,SAF-AはAAA+ドメインによるATPとの結合およびRGGドメインによるクロマチン結合RNAとの結合によりオリゴマーを形成し,また,ATPの加水分解あるいはクロマチン結合RNAの解離により単量体化した.さらに,SAF-Aのオリゴマーの形成は染色体の高次構造を脱凝縮させるために必須であり,逆に,SAF-Aの欠損,あるいは,オリゴマーを形成できない変異型のSAF-Aは染色体の不安定化やDNA損傷への応答機構の活性化をひき起こした.これらの結果から,SAF-Aとクロマチン結合RNAとがおりなす転写に呼応したダイナミックな複合体が染色体の高次構造を制御し,その結果,ゲノムの不安定性からの保護に重要な役割をはたすことが明らかにされた.
クロマチン構造は遺伝子の発現およびゲノムの安定性の維持にきわめて重要なはたらきをもつため,クロマチン構造およびその制御機構の理解はきわめて重要な課題である.これまでに,筆者らの研究グループは,間期の染色体の高次構造において,遺伝子の密度の高いゲノム領域は脱凝縮しており,一方,遺伝子の密度の低いゲノム領域は凝縮していること,そして,転写およびトポイソメラーゼの活性がクロマチンのスーパーコイル構造を制御し,それらがさらに染色体の高次構造に影響をおよぼすことを見い出した1,2).しかし,間期における染色体の高次構造の維持および制御の分子機構についてはほとんど理解されていないのが現状である.かつて,不溶性のタンパク質とRNAから構成される安定な構造体であるとされる核マトリックスが,間期における染色体の構造や染色体テリトリーの維持,遺伝子の発現の制御,核における反応の足場などに重要な役割をはたすというモデルが提唱された3).しかし,生きた細胞において核マトリックスの存在が確認されていないこと,間期における染色体のふるまいがダイナミックであることから,染色体の高次構造の理解に新たな着想が必要とされている.
以前より,クロマチンと結合するRNAのクラスとしてクロマチン結合RNA(chromatin-associated RNA:caRNA)が知られている4).近年,クロマチン結合RNAは間期において染色体テリトリーと相互作用すること,また,その欠損により染色体の凝縮がひき起こされるという報告がなされた5).さらに,クロマチン結合RNAが一過的にクロマチンと結合しクロマチン構造をダイナミックに制御するといったモデルが新たに提唱された6).しかし,クロマチン結合RNAがいかに染色体の高次構造に寄与するかという問いについて具体的な答えはいまだない.
そこで,筆者らは,以前に核マトリックスの構成タンパク質として発見されたSAF-Aに着目した.SAF-AはhnRNP Uとしても知られ,核に豊富に存在し,これまで,スプライシング,DNA損傷部位への蓄積,不活性化X染色体におけるXIST RNAの安定化など,核におけるさまざまな機能にかかわることが報告されているが,SAF-Aの分子機能に関しての知見はとぼしい.この研究においては,SAF-Aがクロマチン結合RNAとともに間期における染色体の高次構造を制御することを発見し,その分子機構を明らかにした.
SAF-Aが染色体の構造に寄与するかどうかを調べるため,RNAi法によりSAF-Aをノックダウンし,DNA蛍光in situハイブリダイゼーション法により染色体の高次構造を解析した.その結果,脱凝縮の状態にあった遺伝子の密度の高い領域の染色体の構造が顕著に凝縮し,一方,遺伝子の密度の低い領域に変化はなかった.この結果は,SAF-Aが転写の活性した領域に優先的に分布するという免疫染色法による解析の結果とよく一致した.さらなる解析により,SAF-Aのノックダウンは,約10 Mbにもおよぶヒトの第11染色体の遺伝子の密度の高い領域や,遺伝子の量の多いヒトの第19染色体の領域において顕著な凝縮をひき起こした.以上の結果から,SAF-Aは遺伝子の密度の高い染色体の領域の構造を100 kbから染色体の全体までのスケールで制御することがわかった.
これまでに筆者らの研究グループは,転写もまた間期において染色体の高次構造を脱凝縮させることを報告している7).そこで,SAF-Aと転写との関係について調べたところ,SAF-Aをノックダウンした条件において転写を阻害してもさらなる染色体の構造の凝縮はみられなかったことから,SAF-Aと転写は染色体の構造を協調的に制御することが示唆された.さらに,転写の阻害によりSAF-Aが核の画分から可溶化したこと,一方,SAF-Aのノックダウンは転写に影響をおよぼさなかったことから,SAF-Aは転写に依存的に染色体の構造を制御すると結論づけられた.
SAF-Aがどのように染色体の構造の制御に寄与するか調べるため,SAF-Aの構造に着目した.SAF-Aは,DNA結合能をもつと考えられているSAPドメイン,機能未知のSPRYドメイン,AAA+ドメイン,RNA結合能をもつと考えられているRGGドメインの4つのドメインから構成される.SAF-AのAAA+ドメインの3次元ホモロジーモデリングにより高度に保存されたWalker AモチーフおよびWalker Bモチーフをもつことが明らかにされ,それらはそれぞれATPとの結合およびATPの加水分解に必須であることから,AAA+ドメインはATPase活性をもつことが予想された.そこで,AAA+ドメインの組換え体を用いて解析したところ,AAA+ドメインはATPase活性をもつことが確認された.さらに,AAA+ドメインおよびRGGドメインをもつ組換え体を用いて解析したところ,RNAの添加によりATPase活性は上昇し,逆に,RNAの分解によりATPase活性は低下した.これらの結果から,SAF-AはRNAに依存的なATPase活性をもつことが示唆された.
SAF-AのAAA+ドメインは典型的なAAA+ドメインにはみられないαヘリックスを1つ余分にもつ.このαヘリックスは,ほかのモチーフの配置からDnaAやORCファミリーにおいて同定されているISMモチーフと相同なモチーフと予想された8).典型的なAAA+ドメインはそのドメインどうし数個が相互作用しリング型の複合体を形成することが知られているが,ISMモチーフは結合した相手のAAA+ドメインに作用し,フラットなリング型ではなく,らせん型のオリゴマーの形成に寄与することが知られている8).蛍光分光光度計を用いた解析により,ATPとの結合がAAA+ドメインに構造の変化をひき起こすことが判明しため,SAF-AはATPとの結合によりオリゴマーを形成すると予想された.
SAF-Aがオリゴマーを形成するかどうか調べるため,タンパク質架橋剤を用いてタンパク質相互作用を固定しウェスタンブロット法により解析したところ,SAF-Aの分子量に対応したはしご状の規則的なバンドのパターンが検出され,SAF-Aがオリゴマーを形成することが示唆された.さらに,SAF-Aの断片を用いた同様の解析から,SAF-Aのオリゴマーの形成にはAAA+ドメインおよびRGGドメインの両方が必要であることがわかった.この結果は,同様の解析において,AAA+ドメインにおける点変異がSAF-Aのオリゴマーの形成に影響をおよぼしたこと,転写の阻害によりSAF-Aのオリゴマーが減少したこととよく一致した.くわえて,細胞から精製したSAF-Aを用いた解析により,RNAは単量体のSAF-AではなくSAF-Aのオリゴマーと結合することが明らかにされた.
SAF-Aのオリゴマー化および単量体化の分子機構について解析した.単量体のSAF-AにRNAをくわえるとSAF-Aのオリゴマーが形成されたこと,また,そのオリゴマーの形成はApyraseにより反応溶液に存在するATPやADPを分解するとみられなくなったことから,SAF-Aのオリゴマーの形成にはRNAおよびATPが必要であることがわかった.一方,SAF-Aのオリゴマーに対しRNase AあるいはApyraseを処理するとSAF-Aは単量体にもどった.これらのことをあわせると,SAF-AはRNAおよびATPとの結合によりオリゴマーを形成し,ATPの加水分解あるいはRNAの解離により単量体にもどる,といったサイクルでオリゴマーを形成するというモデルが考えられた(図1).
実際に,核におけるSAF-Aのオリゴマーの形成を証明するため,FLIM-FRET(fluorescence lifetime imaging microscopy-fluorescence resonance energy transfer)法および超解像度顕微鏡などのイメージングの手法を用いて解析したところ,核には転写に依存的なSAF-Aどうしの相互作用が存在すること,さらに,ATPと結合できない変異型のSAF-Aどうしには相互作用はみられず,ATPを加水分解できない変異型のSAF-Aはより大きなオリゴマーを形成することがわかった.これらの結果から,核においてSAF-Aのオリゴマーの形成のサイクルが存在することが強く支持された(図1).
野生型のSAF-Aを変異型のSAF-Aと置き換えた相補実験において,ATPと結合できない変異型のSAF-AはSAF-Aのノックダウンにより観察された染色体の構造の凝縮の表現型を相補できず,それに対し,ATPの加水分解のできない変異型のSAF-Aはその表現型を相補した.さらに,転写の阻害を組み合わせて染色体の構造の凝縮を促進した条件においても,ATPの加水分解のできない変異型のSAF-Aを発現させた細胞において染色体の凝縮はみられなかった.これらの結果から,SAF-AのATPとの結合を介したオリゴマーの形成は染色体の脱凝縮に,ATPの加水分解を介した単量体化は染色体の凝縮に必要であると結論づけられた.
SAF-Aがクロマチンとどのように相互作用するのか調べた.RNaseの処理によりすべてのSAF-Aが核の画分から可溶化したことから,SAF-Aはクロマチン結合RNAを介してクロマチンと結合することが予想された.ヒトの第3染色体を安定に1つだけもつハムスターハイブリット細胞を用いて,ヒトの染色体に由来するクロマチン結合RNAを特異的に解析したところ,SAF-Aの欠損はクロマチン結合RNAの局在には影響をおよぼさなかったが,転写を阻害した条件においてクロマチン結合RNAは顕著に減少し染色体の構造の凝縮がみられた.さらに,RNAとの結合に必要なRGGドメインを欠失したSAF-Aは染色体の構造の凝縮の表現型を相補しなかったことから,SAF-Aのオリゴマーはクロマチン結合RNAを介して染色体の構造を制御すると結論づけられた.新規に合成されたRNAを標識してクロマチン結合RNAの挙動を観察したところ,クロマチン結合RNAの半減期は約1時間であることが判明し,クロマチン結合RNAは非常に短時間でターンオーバーすることがわかった.これらの結果をあわせて,SAF-Aはクロマチン結合RNAとの相互作用を介し染色体の構造をダイナミックに制御することが明らかにされた.
筆者らは,今回の研究において,SAF-AはATPase活性およびクロマチン結合RNAとの結合を介してオリゴマー化し,染色体の構造を制御することを明らかにした.この発見は,以前に提唱された核マトリックスモデルとは異なり,SAF-Aを中心とした転写に呼応するダイナミックな“クロマチンメッシュ”により染色体の高次構造が制御されるというまったく新しい着想をあたえるものである(図2).
また,マウスにおいてSAF-Aは胚発生に必要であること9),ヒトにおいてSAF-Aのハプロ不全により神経発達異常がもたらされること10) が知られている.実際に,SAF-Aのオリゴマーの形成は間期における染色体の安定性に必要であること,SAF-Aの欠損によりDNA損傷に対する応答機構が活性化することも明らかにされた.今回の研究は,染色体の構造の維持は染色体の安定性の維持に必須であることを直接的に示すものである.
略歴:2011年 北海道大学大学院先端生命科学院にて博士号取得,同年 同 ポスドクを経て,2013年より英国Edinburgh大学 博士研究員.
研究テーマ:染色体の高次構造の分子機構.
抱負:既存の知見にとらわれず,独自性のある実験や研究を日々行っていきたい.
Nick Gilbert
英国Edinburgh大学 教授.
研究室URL:http://www.chromatinlab.org
© 2017 野澤竜介・Nick Gilbert Licensed under CC 表示 2.1 日本
(英国Edinburgh大学Institute of Genetics and Molecular Medicine)
email:野澤竜介
DOI: 10.7875/first.author.2017.068
SAF-A regulates interphase chromosome structure through oligomerization with chromatin-associated RNAs.
Ryu-Suke Nozawa, Lora Boteva, Dinesh C. Soares, Catherine Naughton, Alison R. Dun, Adam Buckle, Bernard Ramsahoye, Peter C. Bruton, Rebecca S. Saleeb, Maria Arnedo, Bill Hill, Rory R. Duncan, Sutherland K. Maciver, Nick Gilbert
Cell, 169, 1214-1227.e18 (2017)
要 約
真核生物においてDNAはクロマチン構造をとり密に折りたたまれて核に収納されている.しかし,その染色体の構造を維持あるいは制御する分子機構の全容はいまだ明らかにされていない.筆者らは,SAF-Aがクロマチン結合RNAと結合し,ヒトの間期における染色体の高次構造を転写に依存的に制御することを見い出した.その分子機構についてくわしく調べたところ,SAF-AはAAA+ドメインによるATPとの結合およびRGGドメインによるクロマチン結合RNAとの結合によりオリゴマーを形成し,また,ATPの加水分解あるいはクロマチン結合RNAの解離により単量体化した.さらに,SAF-Aのオリゴマーの形成は染色体の高次構造を脱凝縮させるために必須であり,逆に,SAF-Aの欠損,あるいは,オリゴマーを形成できない変異型のSAF-Aは染色体の不安定化やDNA損傷への応答機構の活性化をひき起こした.これらの結果から,SAF-Aとクロマチン結合RNAとがおりなす転写に呼応したダイナミックな複合体が染色体の高次構造を制御し,その結果,ゲノムの不安定性からの保護に重要な役割をはたすことが明らかにされた.
はじめに
クロマチン構造は遺伝子の発現およびゲノムの安定性の維持にきわめて重要なはたらきをもつため,クロマチン構造およびその制御機構の理解はきわめて重要な課題である.これまでに,筆者らの研究グループは,間期の染色体の高次構造において,遺伝子の密度の高いゲノム領域は脱凝縮しており,一方,遺伝子の密度の低いゲノム領域は凝縮していること,そして,転写およびトポイソメラーゼの活性がクロマチンのスーパーコイル構造を制御し,それらがさらに染色体の高次構造に影響をおよぼすことを見い出した1,2).しかし,間期における染色体の高次構造の維持および制御の分子機構についてはほとんど理解されていないのが現状である.かつて,不溶性のタンパク質とRNAから構成される安定な構造体であるとされる核マトリックスが,間期における染色体の構造や染色体テリトリーの維持,遺伝子の発現の制御,核における反応の足場などに重要な役割をはたすというモデルが提唱された3).しかし,生きた細胞において核マトリックスの存在が確認されていないこと,間期における染色体のふるまいがダイナミックであることから,染色体の高次構造の理解に新たな着想が必要とされている.
以前より,クロマチンと結合するRNAのクラスとしてクロマチン結合RNA(chromatin-associated RNA:caRNA)が知られている4).近年,クロマチン結合RNAは間期において染色体テリトリーと相互作用すること,また,その欠損により染色体の凝縮がひき起こされるという報告がなされた5).さらに,クロマチン結合RNAが一過的にクロマチンと結合しクロマチン構造をダイナミックに制御するといったモデルが新たに提唱された6).しかし,クロマチン結合RNAがいかに染色体の高次構造に寄与するかという問いについて具体的な答えはいまだない.
そこで,筆者らは,以前に核マトリックスの構成タンパク質として発見されたSAF-Aに着目した.SAF-AはhnRNP Uとしても知られ,核に豊富に存在し,これまで,スプライシング,DNA損傷部位への蓄積,不活性化X染色体におけるXIST RNAの安定化など,核におけるさまざまな機能にかかわることが報告されているが,SAF-Aの分子機能に関しての知見はとぼしい.この研究においては,SAF-Aがクロマチン結合RNAとともに間期における染色体の高次構造を制御することを発見し,その分子機構を明らかにした.
1.SAF-Aは間期における染色体の構造を転写に依存的に制御する
SAF-Aが染色体の構造に寄与するかどうかを調べるため,RNAi法によりSAF-Aをノックダウンし,DNA蛍光in situハイブリダイゼーション法により染色体の高次構造を解析した.その結果,脱凝縮の状態にあった遺伝子の密度の高い領域の染色体の構造が顕著に凝縮し,一方,遺伝子の密度の低い領域に変化はなかった.この結果は,SAF-Aが転写の活性した領域に優先的に分布するという免疫染色法による解析の結果とよく一致した.さらなる解析により,SAF-Aのノックダウンは,約10 Mbにもおよぶヒトの第11染色体の遺伝子の密度の高い領域や,遺伝子の量の多いヒトの第19染色体の領域において顕著な凝縮をひき起こした.以上の結果から,SAF-Aは遺伝子の密度の高い染色体の領域の構造を100 kbから染色体の全体までのスケールで制御することがわかった.
これまでに筆者らの研究グループは,転写もまた間期において染色体の高次構造を脱凝縮させることを報告している7).そこで,SAF-Aと転写との関係について調べたところ,SAF-Aをノックダウンした条件において転写を阻害してもさらなる染色体の構造の凝縮はみられなかったことから,SAF-Aと転写は染色体の構造を協調的に制御することが示唆された.さらに,転写の阻害によりSAF-Aが核の画分から可溶化したこと,一方,SAF-Aのノックダウンは転写に影響をおよぼさなかったことから,SAF-Aは転写に依存的に染色体の構造を制御すると結論づけられた.
2.SAF-AのAAA+ドメインはRNAに依存的なATPase活性をもちATPとの結合により構造が変化する
SAF-Aがどのように染色体の構造の制御に寄与するか調べるため,SAF-Aの構造に着目した.SAF-Aは,DNA結合能をもつと考えられているSAPドメイン,機能未知のSPRYドメイン,AAA+ドメイン,RNA結合能をもつと考えられているRGGドメインの4つのドメインから構成される.SAF-AのAAA+ドメインの3次元ホモロジーモデリングにより高度に保存されたWalker AモチーフおよびWalker Bモチーフをもつことが明らかにされ,それらはそれぞれATPとの結合およびATPの加水分解に必須であることから,AAA+ドメインはATPase活性をもつことが予想された.そこで,AAA+ドメインの組換え体を用いて解析したところ,AAA+ドメインはATPase活性をもつことが確認された.さらに,AAA+ドメインおよびRGGドメインをもつ組換え体を用いて解析したところ,RNAの添加によりATPase活性は上昇し,逆に,RNAの分解によりATPase活性は低下した.これらの結果から,SAF-AはRNAに依存的なATPase活性をもつことが示唆された.
SAF-AのAAA+ドメインは典型的なAAA+ドメインにはみられないαヘリックスを1つ余分にもつ.このαヘリックスは,ほかのモチーフの配置からDnaAやORCファミリーにおいて同定されているISMモチーフと相同なモチーフと予想された8).典型的なAAA+ドメインはそのドメインどうし数個が相互作用しリング型の複合体を形成することが知られているが,ISMモチーフは結合した相手のAAA+ドメインに作用し,フラットなリング型ではなく,らせん型のオリゴマーの形成に寄与することが知られている8).蛍光分光光度計を用いた解析により,ATPとの結合がAAA+ドメインに構造の変化をひき起こすことが判明しため,SAF-AはATPとの結合によりオリゴマーを形成すると予想された.
3.SAF-AはATPおよびRNAに依存的にオリゴマーを形成する
SAF-Aがオリゴマーを形成するかどうか調べるため,タンパク質架橋剤を用いてタンパク質相互作用を固定しウェスタンブロット法により解析したところ,SAF-Aの分子量に対応したはしご状の規則的なバンドのパターンが検出され,SAF-Aがオリゴマーを形成することが示唆された.さらに,SAF-Aの断片を用いた同様の解析から,SAF-Aのオリゴマーの形成にはAAA+ドメインおよびRGGドメインの両方が必要であることがわかった.この結果は,同様の解析において,AAA+ドメインにおける点変異がSAF-Aのオリゴマーの形成に影響をおよぼしたこと,転写の阻害によりSAF-Aのオリゴマーが減少したこととよく一致した.くわえて,細胞から精製したSAF-Aを用いた解析により,RNAは単量体のSAF-AではなくSAF-Aのオリゴマーと結合することが明らかにされた.
SAF-Aのオリゴマー化および単量体化の分子機構について解析した.単量体のSAF-AにRNAをくわえるとSAF-Aのオリゴマーが形成されたこと,また,そのオリゴマーの形成はApyraseにより反応溶液に存在するATPやADPを分解するとみられなくなったことから,SAF-Aのオリゴマーの形成にはRNAおよびATPが必要であることがわかった.一方,SAF-Aのオリゴマーに対しRNase AあるいはApyraseを処理するとSAF-Aは単量体にもどった.これらのことをあわせると,SAF-AはRNAおよびATPとの結合によりオリゴマーを形成し,ATPの加水分解あるいはRNAの解離により単量体にもどる,といったサイクルでオリゴマーを形成するというモデルが考えられた(図1).
実際に,核におけるSAF-Aのオリゴマーの形成を証明するため,FLIM-FRET(fluorescence lifetime imaging microscopy-fluorescence resonance energy transfer)法および超解像度顕微鏡などのイメージングの手法を用いて解析したところ,核には転写に依存的なSAF-Aどうしの相互作用が存在すること,さらに,ATPと結合できない変異型のSAF-Aどうしには相互作用はみられず,ATPを加水分解できない変異型のSAF-Aはより大きなオリゴマーを形成することがわかった.これらの結果から,核においてSAF-Aのオリゴマーの形成のサイクルが存在することが強く支持された(図1).
4.SAF-Aのオリゴマーはクロマチン結合RNAを介して染色体の高次構造を制御する
野生型のSAF-Aを変異型のSAF-Aと置き換えた相補実験において,ATPと結合できない変異型のSAF-AはSAF-Aのノックダウンにより観察された染色体の構造の凝縮の表現型を相補できず,それに対し,ATPの加水分解のできない変異型のSAF-Aはその表現型を相補した.さらに,転写の阻害を組み合わせて染色体の構造の凝縮を促進した条件においても,ATPの加水分解のできない変異型のSAF-Aを発現させた細胞において染色体の凝縮はみられなかった.これらの結果から,SAF-AのATPとの結合を介したオリゴマーの形成は染色体の脱凝縮に,ATPの加水分解を介した単量体化は染色体の凝縮に必要であると結論づけられた.
SAF-Aがクロマチンとどのように相互作用するのか調べた.RNaseの処理によりすべてのSAF-Aが核の画分から可溶化したことから,SAF-Aはクロマチン結合RNAを介してクロマチンと結合することが予想された.ヒトの第3染色体を安定に1つだけもつハムスターハイブリット細胞を用いて,ヒトの染色体に由来するクロマチン結合RNAを特異的に解析したところ,SAF-Aの欠損はクロマチン結合RNAの局在には影響をおよぼさなかったが,転写を阻害した条件においてクロマチン結合RNAは顕著に減少し染色体の構造の凝縮がみられた.さらに,RNAとの結合に必要なRGGドメインを欠失したSAF-Aは染色体の構造の凝縮の表現型を相補しなかったことから,SAF-Aのオリゴマーはクロマチン結合RNAを介して染色体の構造を制御すると結論づけられた.新規に合成されたRNAを標識してクロマチン結合RNAの挙動を観察したところ,クロマチン結合RNAの半減期は約1時間であることが判明し,クロマチン結合RNAは非常に短時間でターンオーバーすることがわかった.これらの結果をあわせて,SAF-Aはクロマチン結合RNAとの相互作用を介し染色体の構造をダイナミックに制御することが明らかにされた.
おわりに
筆者らは,今回の研究において,SAF-AはATPase活性およびクロマチン結合RNAとの結合を介してオリゴマー化し,染色体の構造を制御することを明らかにした.この発見は,以前に提唱された核マトリックスモデルとは異なり,SAF-Aを中心とした転写に呼応するダイナミックな“クロマチンメッシュ”により染色体の高次構造が制御されるというまったく新しい着想をあたえるものである(図2).
また,マウスにおいてSAF-Aは胚発生に必要であること9),ヒトにおいてSAF-Aのハプロ不全により神経発達異常がもたらされること10) が知られている.実際に,SAF-Aのオリゴマーの形成は間期における染色体の安定性に必要であること,SAF-Aの欠損によりDNA損傷に対する応答機構が活性化することも明らかにされた.今回の研究は,染色体の構造の維持は染色体の安定性の維持に必須であることを直接的に示すものである.
文 献
- Gilbert, N., Boyle, S., Fiegler, H. et al.: Chromatin architecture of the human genome: gene-rich domains are enriched in open chromatin fibers. Cell, 118, 555-566 (2004)[PubMed]
- Naughton, C., Avlonitis, N., Corless, S. et al.: Transcription forms and remodels supercoiling domains unfolding large-scale chromatin structures. Nat. Struct. Mol. Biol., 20, 387-395 (2013)[PubMed]
- Berezney, R. & Coffey, D. S.: Identification of a nuclear protein matrix. Biochem. Biophys. Res. Commun., 60, 1410-1417 (1974)[PubMed]
- Holmes, D. S., Mayfield, J. E., Sander, G. et al.: Chromosomal RNA: its properties. Science, 177, 72-74 (1972)[PubMed]
- Hall, L. L., Carone, D. M., Gomez, A. V. et al.: Stable C0T-1 repeat RNA is abundant and is associated with euchromatic interphase chromosomes. Cell, 156, 907-919 (2014)[PubMed]
- Mele, M. & Rinn, J. L.: ‘‘Cat’s Cradling’’ the 3D genome by the act of lncRNA transcription. Mol. Cell, 62, 657-664 (2016)[PubMed]
- Naughton, C., Sproul, D., Hamilton, C. et al.: Analysis of active and inactive X chromosome architecture reveals the independent organization of 30 nm and large-scale chromatin structures. Mol. Cell, 40, 397-409 (2010)[PubMed]
- Erzberger, J. P. & Berger, J. M.: Evolutionary relationships and structural mechanisms of AAA+ proteins. Annu. Rev. Biophys. Biomol. Struct., 35, 93-114 (2006)[PubMed]
- Roshon, M. J. & Ruley, H. E.: Hypomorphic mutation in hnRNP U results in post-implantation lethality. Transgenic Res., 14, 179-192 (2005)[PubMed]
- Deciphering Developmental Disorders Study: Large-scale discovery of novel genetic causes of developmental disorders. Nature, 519, 223-228 (2015)[PubMed]
活用したデータベースにかかわるキーワードと統合TVへのリンク
著者プロフィール
略歴:2011年 北海道大学大学院先端生命科学院にて博士号取得,同年 同 ポスドクを経て,2013年より英国Edinburgh大学 博士研究員.
研究テーマ:染色体の高次構造の分子機構.
抱負:既存の知見にとらわれず,独自性のある実験や研究を日々行っていきたい.
Nick Gilbert
英国Edinburgh大学 教授.
研究室URL:http://www.chromatinlab.org
© 2017 野澤竜介・Nick Gilbert Licensed under CC 表示 2.1 日本